noticias
Desarrollan tambaquis triploides que crecen más rápido
Los científicos de Embrapa Pesca e Aquicultura (TO) estudian una técnica capaz de hacer que el tambaqui (Colossoma macropomum) crezca aproximadamente un 20% más grande …
Read moreColaboración UACh, INCAR e Infood Protein profundizará en la investigación del uso de harina de insecto en dietas de peces
El laboratorio de inmunología y estrés de organismos acuáticos de la Universidad Austral de Chile (UACh), liderado por el Investigador Asociado de la línea “Salud …
Read moreBenchmark Genetics Chile recibe importantes visitas nacionales e internacionales
Puerto Montt, Chile – Una semana con variadas actividades en torno a la feria internacional AquaSur 2024 está cerrando Benchmark Genetics Chile. La casa genética, …
Read moremercado
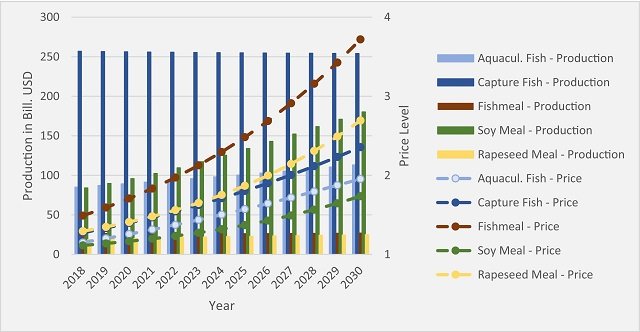
el acuicultor

Acuicultura del Rodaballo Scophthalmus maximus
El rodaballo (Scophthalmus maximus), o turbot, es un pez plano de gran sabor y valor nutricional. Habita en aguas templadas del Atlántico y el Mediterráneo, …